
Денис Владимирович Соломатин
mixOmics для гуманитариев
Введение
«Живые смыслы не оцифровать», тем не менее, реалии цифровой эпохи таковы, что всё настойчивее стремимся вникнуть в тайны мироздания пользуясь предоставляемым математикой инструментарием и описать увиденное на языке цифр. Само по себе это не хорошо и не плохо, не стоит лишь забывать и об эмоциональной, чувственной составляющей жизни человека. В связи со сказанным на передний план выходят различные «омики», изучающие то всеобъемлющее, что буквально создаёт нас, формируя основу нашей жизни. В частности, например: геном – как совокупность данных обо всех наших генах; транскриптом – постоянно меняющийся набор считываемых из генома данных; протеом – все производимые нашим организмом белки; эпигеном – условия, в которых живёт организм, формирующие своеобразный регуляторный уровень над генами; микробиом – бактерии, с которыми мы живём; метагеном – совокупный геном сообщества организмов, живущих вместе; коннектом – совокупность нервных связей живого организма; социом – как совокупность социальных связей индивида. Созданием в определённом смысле этого слова новых членов общества занимается и система образования, именно поэтому на наш взгляд оказывается уместным в ходе статистической обработки педагогической информации использование mixOmics – пакета прикладных программ, функций и процедур R, разрабатываемого и поддерживаемого отделением математики и статистики Мельбурнского университета (Австралия), а также Институтом математики Университета Тулузы (Франция), с передовыми достижениями которых можно ознакомиться на сайте http://mixomics.org
В фундаментальной работе Грабарь М. И., Краснянская К. А. (Применение математической статистики и педагогических исследованиях. Непараметрические методы. М., «Педагогика», 1977. 136 с. с ил. Науч.-исслед. ин-т содержания и методов обучения Акад. пед. наук СССР), на стр.4 констатировали печальный факт: «Любое изложение общей теории проверки статистических гипотез неизбежно должно предполагать у читатели очень серьезную математическую подготовку, каковой, к сожалению, не обладают большинство исследователей-педагогов». С наступлением цифровой эпохи и распространением доступных инструментальных средств статистической обработки информации отмеченный недостаток можно нивелировать и обратить в достоинство. Предполагается, что читатель уже знаком с изложенными в предыдущей части настоящего пособия азами работы R, – языка программирования для статистической обработки данных и работы с графикой, а также свободной программной среды вычислений с открытым исходным кодом в рамках проекта GNU. Поэтому во второй части сконцентрируемся на использовании ключевых функций пакета mixOmics для анализа педагогических данных. Если возникнут какие-либо проблемы с пониманием излагаемого материала, настоятельно рекомендуется вернуться и перечитать предыдущую часть пособия. Выбранный набор инструментов включает в себя многовариантные методы статистического анализа, предпочтение которым отдаётся в зависимости от обрабатываемых или собираемых педагогических данных, например, с целью апробации результатов, дискриминантного анализа, слияния двух или более наборов данных. mixOmics – это набор инструментов R, посвященный исследованию и слиянию различных наборов данных с определенным акцентом на выборе переменных. Пакет в настоящее время включает в себя порядка двадцати многовариантных методов. Первоначально все методы были разработаны для данных «омиков», однако их применение не ограничивается только такими данными. Другие приложения возникают как правило в тех случаях, когда переменные-предикторы (то есть переменные, по значениям которых составляются прогнозы) непрерывны.
В пакете mixOmics, сильный акцент делается на графическое представление, чтобы лучше интерпретировать и понять отношения между различными типами данных визуализируют структуру корреляции как на выборочных значениях, так и на шкале интервалов. А начинается использование рассматриваемого пакета со ввода данных. Напомним блок-схему основного алгоритма статистической обработки педагогических и социальных данных, концептуально выкристаллизовавшегося к концу предыдущей части книги:
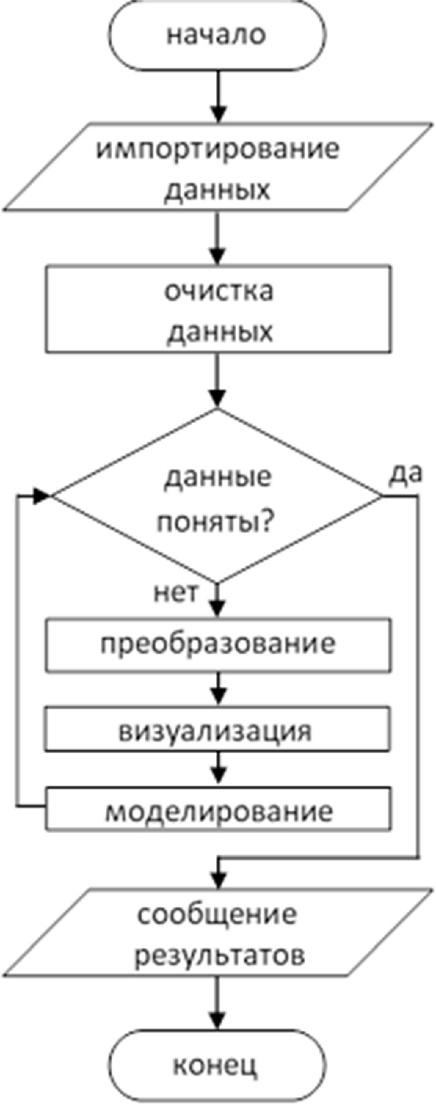
Как видим, обработка начинается со ввода данных, их предварительного импортирования и очистки. К предварительной обработке данных перед анализом данных с помощью mixOmics предъявляются следующие требования:
Различные типы педагогических данных могут быть изучены и интегрированы с mixOmics. Методы могут обрабатывать показатели успеваемости, измеренные в непрерывном масштабе или полученные на основе данных подсчета, которые становятся непрерывными данными после предварительной обработки и нормализации.
Пакет mixOmics не справляется с нормализацией, так как он универсален и охватывает широкий спектр данных. До начала анализа предполагается, что наборы данных были нормализованы с использованием соответствующих методов нормализации педагогических данных и предварительно обработаны, когда это возможно.
В то время как методы mixOmics могут обрабатывать большие массивы данных (несколько десятков тысяч переменных-предикторов), рекомендуется предварительно фильтровать данные до менее чем 10 000 переменных-предикторов на набор данных, например, с помощью медианного абсолютного отклонения, удалив пренебрежимо малые значения в наборах данных или путем удаления предикторов почти нулевой дисперсии. Такой шаг направлен на уменьшение вычислительного времени в процессе настройки параметров.
Методы mixOmics используют разложения матриц. Таким образом, числовая матрица данных или фреймы данных имеют n наблюдений или образцов в строках и p предикторов или переменных в столбцах.
В текущей версии mixOmics, ковариации, которые могут запутать анализ не включены в методы статистического анализа. Рекомендуется корректировать наборы этих ковариаций заранее, используя соответствующие унивариантные или многовариантные методы для удаления информационного шума.
Перечислим теперь основные методологические и теоретические основы, которые необходимо знать, чтобы эффективно применять mixOmics:
• Индивидуальные наблюдения или образцы: экспериментальные группы, на которых собиралась информация, например, обучающиеся, студенты, олимпиадные задания и прочее.
• Переменные, предикторы: считываемые измерения на каждом образце, например, успеваемость, посещаемость, решаемость задач, творческая самореализация и так далее.
• Дисперсия: измеряет уровень распылённости одной переменной. Как правило оценивается дисперсия целых компонентов, а не считываемых переменных. Высокая дисперсия указывает на то, что точки данных очень отличаются от среднего, и друг от друга (разбросаны).
• Ковариация: измеряет прочность взаимосвязи между двумя переменными, то есть являются ли они ковариантами друг друга. Высокое значение ковариации указывает на сильную связь, например, посещаемость и успеваемость у отдельных обучающихся часто различаются примерно одинаково; в общем случае, самые низкие и самые высокие значения коэффициента ковариации не имеют нижнего или верхнего предела.
• Корреляция: нормализованная версия ковариации, значения которой ограничены отрезком от -1 до 1.
• Линейная комбинация: разные переменные могут объединяться в одну путем умножения каждой из них на коэффициент и сложения полученных результатов. Линейная комбинация успеваемость a и посещаемости b может быть 2∙a – 1,5∙b с коэффициентами 2 и -1,5, присвоенных успеваемости и посещаемости соответственно.
• Компонент: искусственная переменная, построенная из линейной комбинации наблюдаемых переменных в данном наборе данных. Переменные коэффициенты оптимально определяются на основе какого-то статистического критерия. Например, в основном компоненте анализа определяются коэффициенты (основного) компонента, с тем чтобы максимизировать дисперсию компонента.
• Нагрузки: переменные коэффициенты, используемые для определения компонента.
• Визуализация образца: представление образцов, проецируемых в небольшом пространстве, охватываемом (определяемом) компонентами. Координаты образцов определяются значениями или вычисленными баллами компонентов.
• Изображение круга корреляции: представление переменных в пространстве, охватываемом компонентами. Каждая переменная координата определяется как корреляция между исходным переменным значением и каждым компонентом. Диаграмма с корреляционным кругом позволяет визуализировать корреляцию между переменными – отрицательную или положительную корреляцию, определяемую косинусом угла между центром круга и каждой переменной точкой, а также вклад каждой переменной в каждый компонент, определяемый абсолютным значением координат по каждому компоненту. Для такого толкования данные должны быть сосредоточены и масштабированы, что подразумевается по умолчанию в большинстве методов, за исключением PCA. Подробная информация об этом наглядном представлении информации будет представлена в соответствующем разделе ниже.
• Неконтролируемый анализ: метод, который не учитывает какие-либо известные группы выборки, является исследовательским. Примерами неконтролируемых методов являются – метод главных компонент (PCA), метод проекций на скрытые структуры (PLS), а также канонический анализ корреляции (CCA).
• Контролируемый анализ: метод включает вектор, указывающий на принадлежность класса в каждой выборки. Цель его состоит в том, чтобы различать выборочные группы и выполнять прогнозирование для класса выборки. Примерами контролируемых методов являются дискриминантный анализ проекций на скрытые компоненты (PLS-DA), анализ интеграции данных для обнаружения маркеров с использованием скрытых компонентов (DIAB), а также многомерный интегративный метод определения воспроизводимых сигнатур в независимых экспериментах на разных платформах (MINT).
Перечень широко используемых методов mixOmics, которые будут подробно описаны в соответствующих главах ниже, за исключением CCA и MINT, можно представить следующей таблицей типов и объема данных, который они могут обрабатывать:
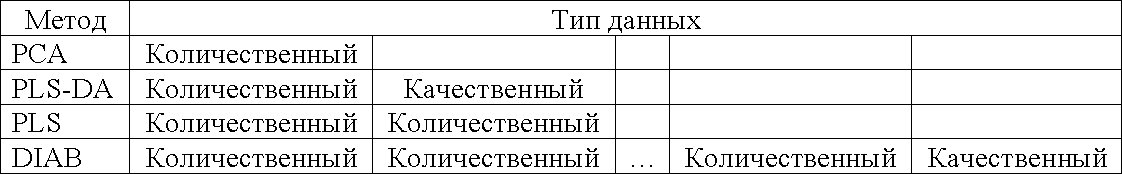
Методы, реализованные в mixOmics, подробно описаны в разных публикациях, обширный список которых постоянно пополняется и может быть найден в открытых источниках.
В следующей таблице приведён список методов mixOmics, наличие разрежения в которой указывает на методы, предполагающие осуществление выбора переменных:
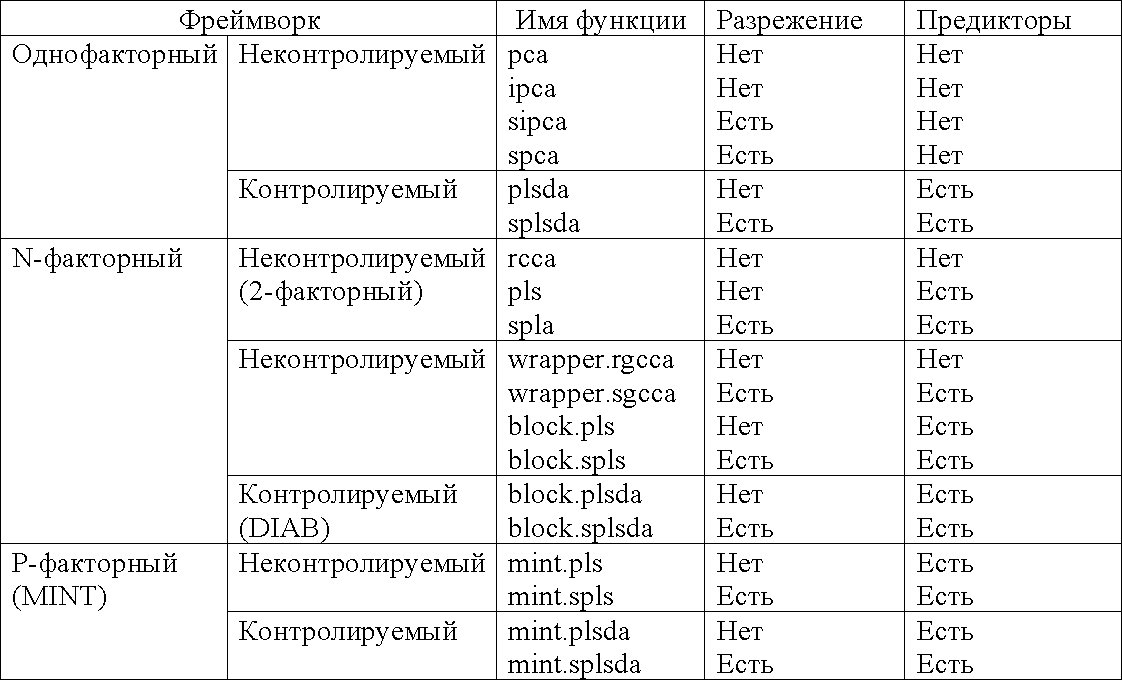
Основные функции и параметры каждого метода сведены в следующей таблице:
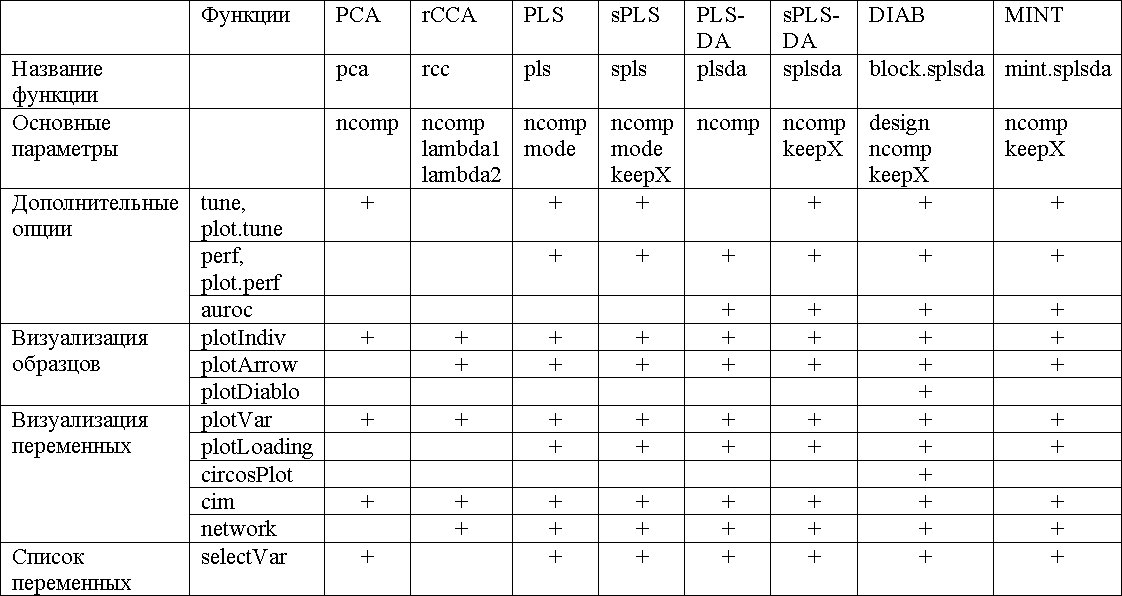
Каждый раздел, посвященный описанию того или иного метода, излагается по следующему плану:
1. Тип педагогического вопроса, на который нужно ответить.
2. Краткое описание иллюстративного набора данных.
3. Принцип метода.
4. Быстрый запуск метода с основными функциями и аргументами.
5. Чтобы идти дальше: настраиваемые опции, дополнительные графические построения и настройки параметров.
6. Вопросы и ответы.
Глава 1. Первые шаги
Как путь в тысячу миль начинается с первого шаг, так и использование любого пакета R начинается с его установки. Во-первых, можно скачать последнюю версию mixOmics от Bioconductor следующей командой:
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("mixOmics")
Кроме того, можно установить последнюю версию пакета с GitHub, но для этого понадобится предварительная установка пакета remotes:
BiocManager::install("remotes")
BiocManager::install("mixOmicsTeam/mixOmics")
Пакет mixOmics напрямую импортирует следующие пакеты: igraph, rgl, ellipse, corpcor, RColorBrewer, plyr, parallel, dplyr, tidyr, reshape2, methods, matrixStats, rARPACK, gridExtra. Если возникнут затруднения при установке пакета rgl, то нужно будет дополнительно установить программное обеспечение X'quartz.
Загрузить установленный пакет можно следующей командой:
library(mixOmics)
Убедитесь, что при загрузке пакета не возникло ошибки, особенно для упомянутой выше библиотеки rgl. В примерах, которые будут приведены далее, используются данные, являющиеся частью пакета mixOmics. Чтобы загрузить свои собственные данные, проверьте установлен ли рабочий каталог, а затем считайте данные из формата .txt или .csv, либо с помощью пункта меню импортирования данных в RStudio, либо через одну из следующих командных строк:
# из файла csv
data <– read.csv("имя_файла.csv", row.names = 1, header = TRUE)
# из файла txt
data <– read.table("имя_файла.txt", header = TRUE)
Для получения более подробной информации о аргументах, используемых для настройки параметров этих функций, введите ?read.csv или ?read.table в консоли R.
Каждый анализ должен выполняться в следующем порядке:
1. Запустите выбранный метод анализа.
2. Выполните графическое представление образцов.
3. Выполните графическое представление переменных.
Затем используйте критическое мышление и дополнительные функции инструментов визуализации, чтобы разобраться в полученных данных. Некоторые из вспомогательных инструментов будут описаны в следующих главах.
Например, для анализа основных компонентов сначала загружаем данные:
My_table <– structure(list(Класс = c("7а", "7а", "7а", "7а", "7а", "7а", "7а", "7а", "7а",
"7а", "7б", "7б", "7б", "7б", "7б", "7б", "7б", "7б", "7б", "7б", "эталон", "отстающий"),
`Фимилия Имя` = c("Иванов Иван", "Петров Петр", "Сидоров Сидор", "Егоров Егор",
"Романов Роман", "Николаев Николай", "Григорьев Григогий", "Викторов Виктор",
"Михайлов Михаил", "Тимуриев Тимур", "Ульянова Ульяна", "Ольгина Ольга",
"Людмилова Людмила", "Дарьева Дарья", "Кристинина Кристина",
"Натальина Наталья", "Глафирова Глафира", "Янина Яна", "Иринова Ирина",
"Валентинова Валентина", "Идеальный ученик", "Другая крайность"), Тема1 = c(5, 5, 5, 5, 5, 5, 5, 5, 5, 5, 5, 5, 5, 5, 5, 5, 5, 5, 5, 5, 5, 1), Тема2 = c(2, 3, 3, 2, 2, 3, 3, 2, 2, 3, 4, 5, 5, 4, 4, 4, 5, 5, 4, 5, 5, 1), Тема3 = c(1, 2, 2, 1, 1, 2, 2, 1, 2, 2, 1, 2, 2, 1, 1, 2, 2, 2, 1, 2, 5, 1), Тема4 = c(4, 5, 5, 4, 4, 4, 5, 5, 5, 4, 5, 5, 4, 4, 5, 5, 4, 4, 5, 4, 5, 1), `Тема 5` = c(1, 2, 2, 2, 1, 2, 1, 1, 2, 2, 1, 2, 2, 1, 1, 2, 2, 1, 2, 5, 5, 1), `№№` = c(1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22)), row.names = c(NA, -22L), class = c("tbl_df", "tbl", "data.frame"))
Затем выполним следующие шаги:
My_result.pca <– pca(My_table) # 1 Запуск выбранного метода анализа
plotIndiv(My_result.pca) # 2 Визуальное представление образцов
plotVar(My_result.pca) # 3 Визуальное представление переменных
Это только первый пример, в дальнейшем появится много вариантов, из которых можно будет выбрать, наиболее соответствующий стоящим перед вами исследовательским задачам статистического анализа. Пакет mixOmics предлагает различные методы представления переменной и широкий выбор функций сбора информации на довольно больших наборах данных.
Сохраним числовые данные из исходной таблицы во вспомогательной переменной:
to.remove <– c('Фимилия Имя', 'Класс', '№№')
X <– My_table[, !colnames(My_table) %in% to.remove]
Следуя примеру выше, методы PCA могут быть применены для выбора первых пяти переменных, тесно связанных с первыми двумя компонентами в PCA. Пользователь определяет количество переменных, выбранных по каждому компоненту, например, здесь выберем пять переменных на каждом из первых двух компонентов командой keepX=c(5,5):
My_result.spca <– spca(X, keepX=c(5,5)) # 1 Запуск выбранного метода анализа
plotIndiv(My_result.spca) # 2 Визуальное представление образцов
plotVar(My_result.spca) # 3 Визуальное представление переменных

Можно заметить, что сократилось количество элементов на круге корреляции. Не останавливайтесь на достигнутом, находясь в начале большого пути. Можно улучшить наглядность представляемых результатов анализа следующим образом: загляните в справочное руководство по каждой из функций используемой в примерах, введя в консоли ?pca, ?plotIndiv, ?sPCA. Для запуска сопутствующих примеров можно использовать функцию example: example(pca), example(plotIndiv), и другие.





